研究项目
病理 CLIP 常见测试方法及数据集分享
 龙瀚林
龙瀚林
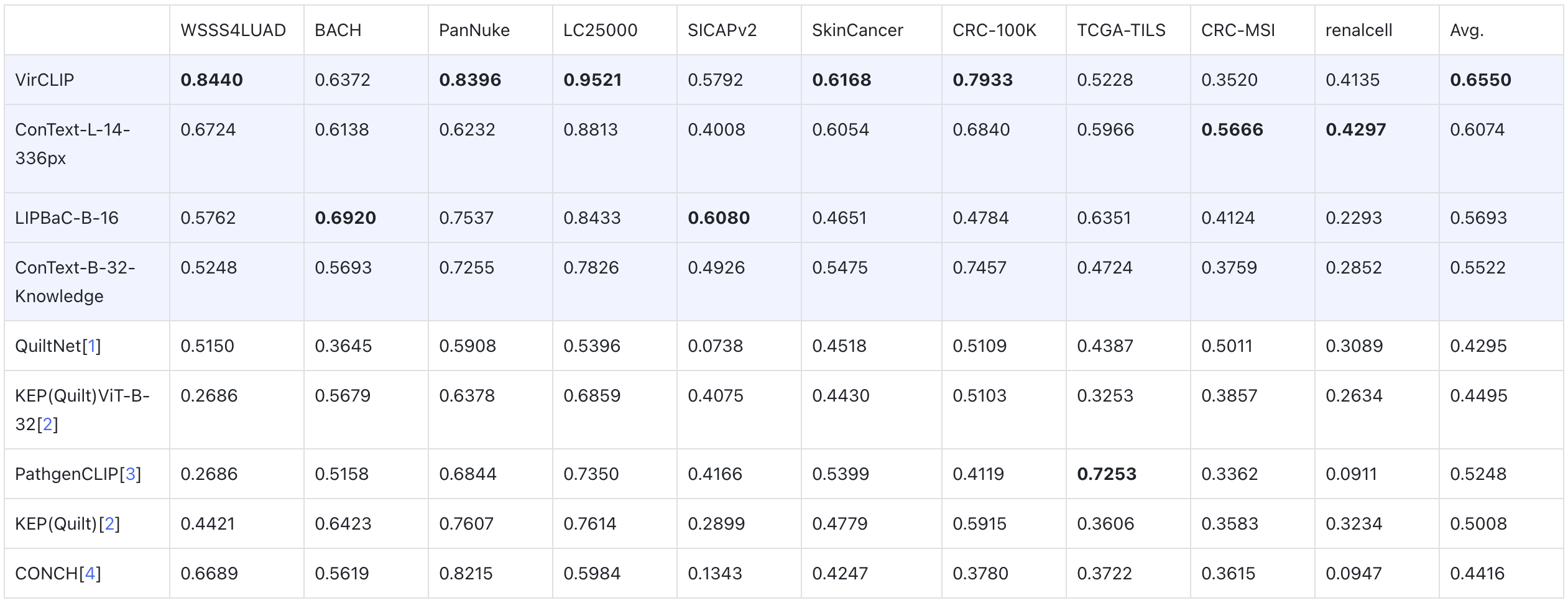
病理多模态数据集中的非病理图像过滤
现有公开病理图文配对数据集(如Quilt-1m)通过截取YouTube视频片段构建,尽管已实施初步过滤策略,但仍存在显著噪声(如非病理图像)。基于不同数据规模与模型架构的分类器训练表明,各类模型在分类性能上存在显著差异。实验验证表明,通过过滤非病理数据构建的优化数据集对大模型进行微调,能够显著提升其在下游任务上的表现。
 戚文瑾
戚文瑾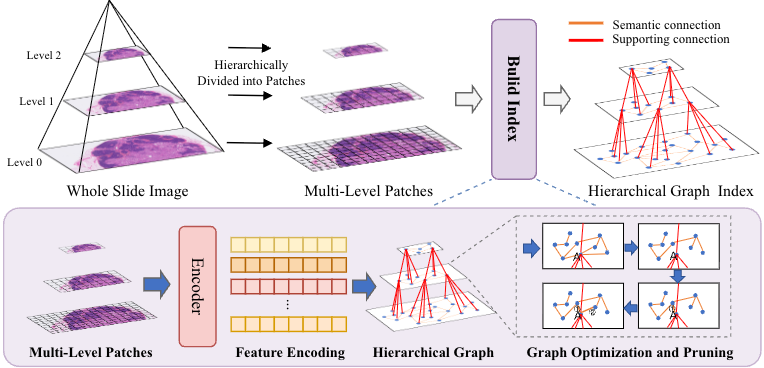
向量检索及GPU加速
本项目提出一种面向全切片病理图像(WSI)的分层检索框架与高效检索方法。该框架通过构建多层级向量索引,保留了病理图像在不同放大倍率下的空间层级结构与语义信息。在检索时,系统利用GPU加速的并行计算流程实现对任意尺寸查询区域的快速响应,并通过拓扑重组算法将匹配的图像补丁聚合成具有诊断意义的连续区域。该方法显著提升了大规模病理图像检索的精度和速度,解决了传统方法在处理多尺度、可变区域查询时的灵活性不足和效率低下的问题。
 司永浩
司永浩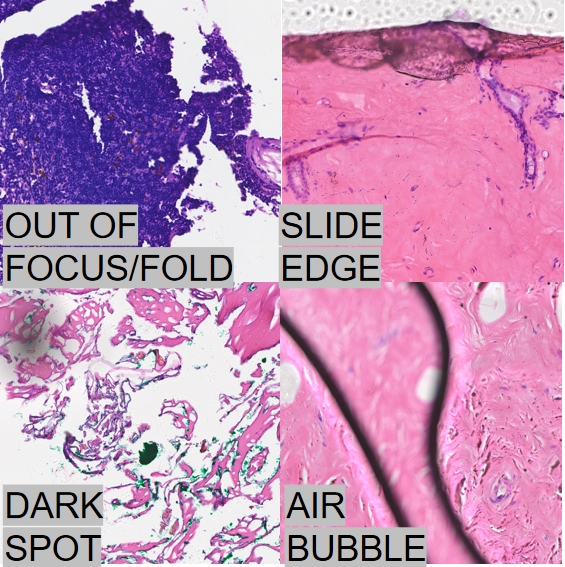
无需训练的病理图像块质量控制方法
病理数字切片的制作流程涉及多个关键环节,其中任一环节的潜在质量问题均可能引发图像失焦、组织重叠等缺陷。这些异常区域会导致病理组织结构信息缺失,严重影响临床诊断的准确性和可靠性。因此,亟需建立一种快速、高效的算法模型,实现问题区域的精准识别与过滤,并深入探究此类低质图像块对病理智能分析模型训练的干扰机制及其影响程度。
 戚文瑾
戚文瑾
乳腺癌中的多模态大模型评估
评估多模态大型语言模型(MLLMs)在乳腺癌任务中的表现。
 黄飞瑜
黄飞瑜
基于CLAM的图像描述生成
基于CLAM的现有能力对WSI图片进行弱监督-少样本的标注。
 郭鹏宇
郭鹏宇
基于知识库增强的病理学CLIP(公共数据集)
基于知识库增强的病理学CLIP致力于解决病理学基础模型在不同病理学领域性能变化的问题。
 龙瀚林
龙瀚林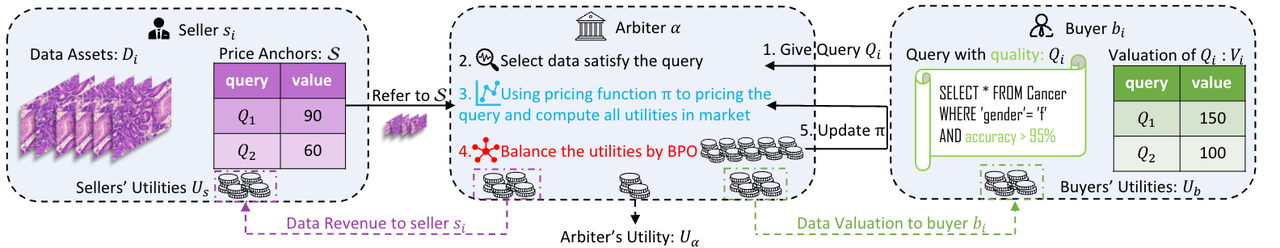
图像描述数据市场演示
我们的研究专注于构建一个全面的基准来评估其性能。
 郭松岳
郭松岳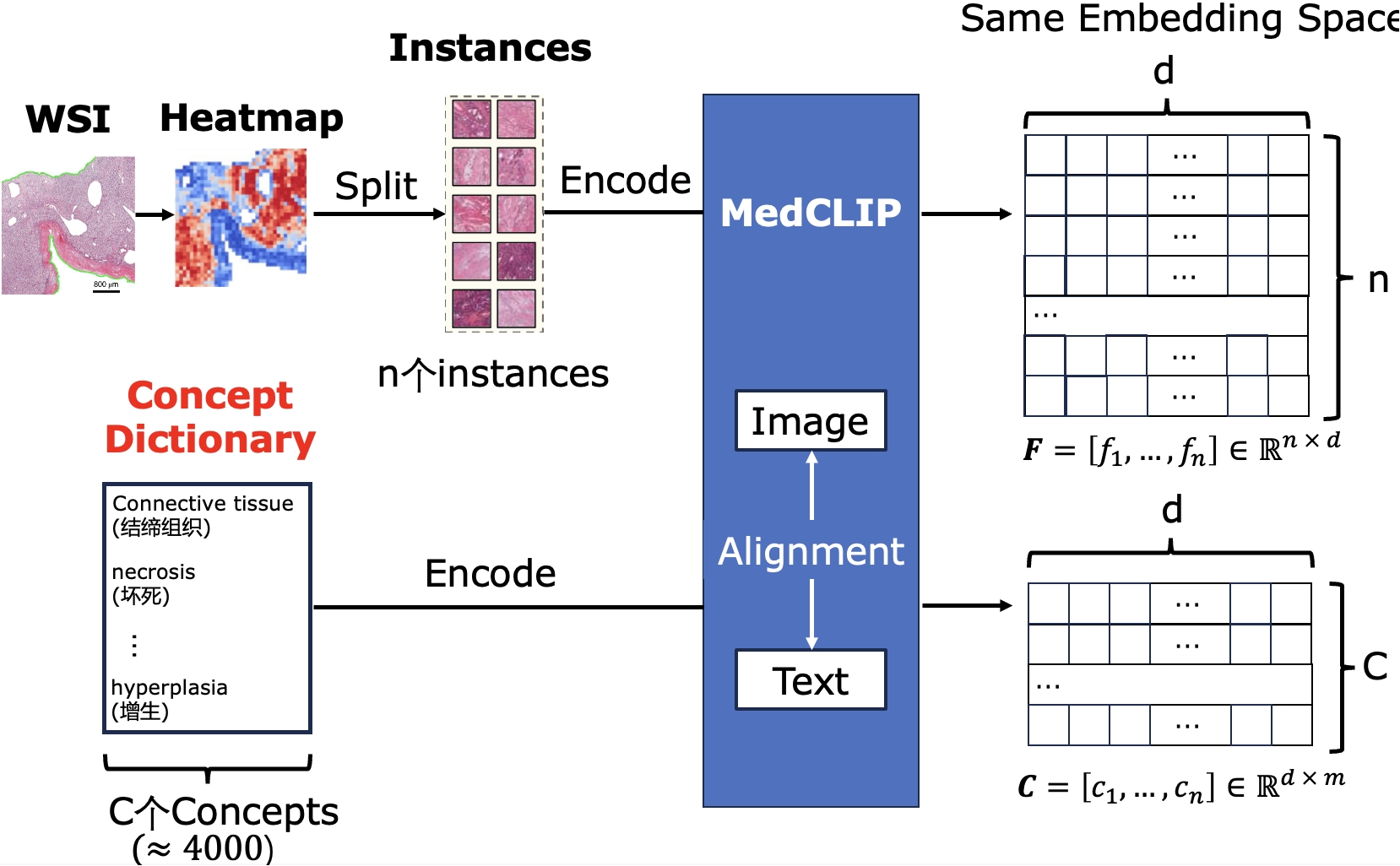
基于多实例学习的病理图文结构化对齐
通过海量病理文本对训练出的病理基础模型提供了强大的病理图文对齐能力。
 郭鹏宇
郭鹏宇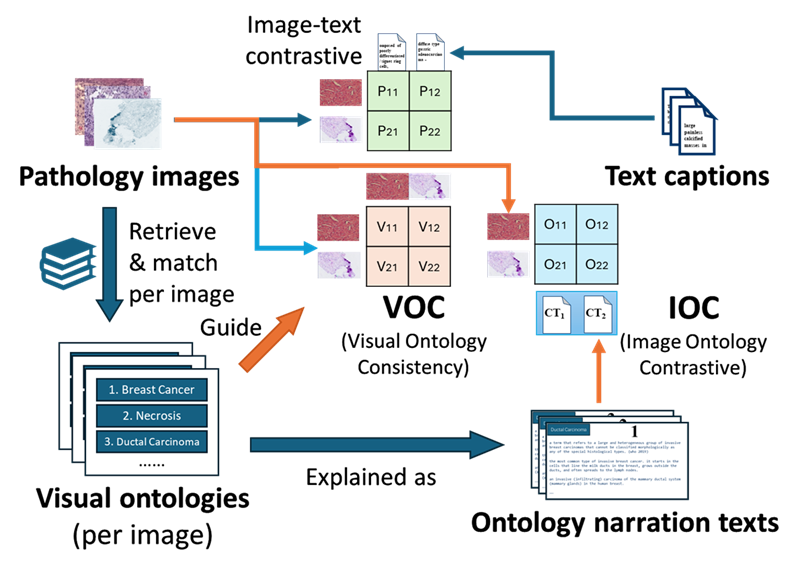
Pivot:用病理知识库增强病理图文对齐
对齐病理图像、病理条目和文本。
 龙瀚林
龙瀚林
RIVL:通过插值法解决在病理图文对齐中遇到的模态缺失问题
对齐病理图像、病理条目和文本。
 龙瀚林
龙瀚林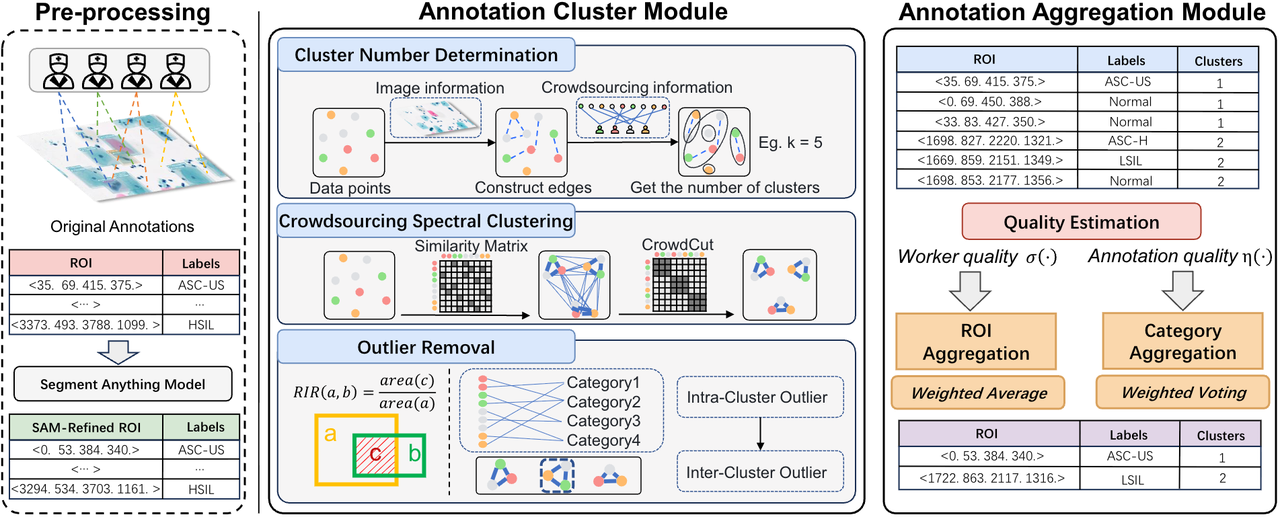
复杂医疗图像标注聚合框架专利
本项目提出一种针对复杂众包标签的聚合方法,该方法融合了计算机视觉基础模型与新型的众包谱聚类技术。系统首先利用视觉基础模型结合图像信息对原始标注进行微调,以突破标注者能力上限;随后,通过一种结合深度优先搜索的迭代式聚类流程(确定簇数-聚类-去离群点),有效地对指向同一实体的复杂标注(如边界框)进行分组;最后,根据标注者和标注结果的质量评估进行加权聚合。该方案显著提升了多目标、多类别复杂标签聚合的准确性,为高质量数据获取提供了新的框架。
 司永浩
司永浩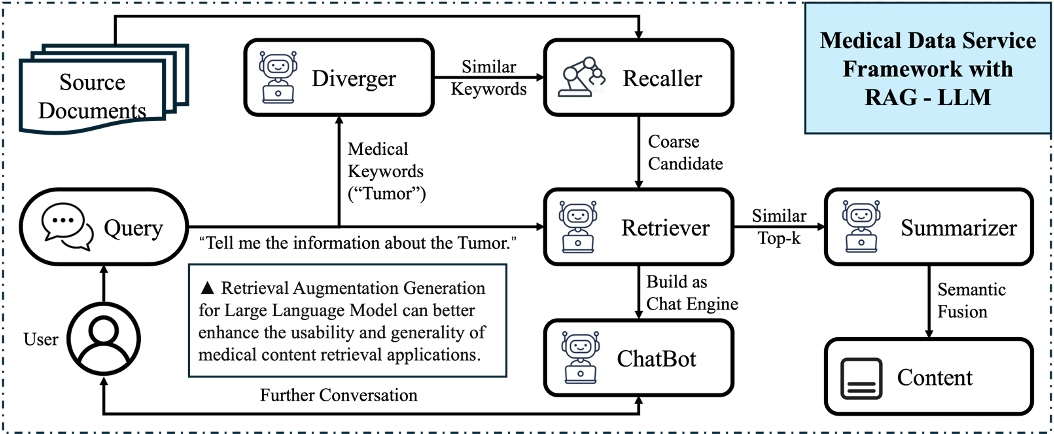
一种问答处理方法、装置、电子设备、存储介质及产品
本项目提出一种结合大语言模型(LLM)与检索增强生成(RAG)的医疗病理知识检索方案。系统通过医疗知识图谱扩展查询语义,并基于相似度索引筛选相关内容,从而提升检索的准确性与专业性。最终,大语言模型对结果进行总结与对话生成,实现智能、可靠的医学知识检索与辅助分析。
 粟日
粟日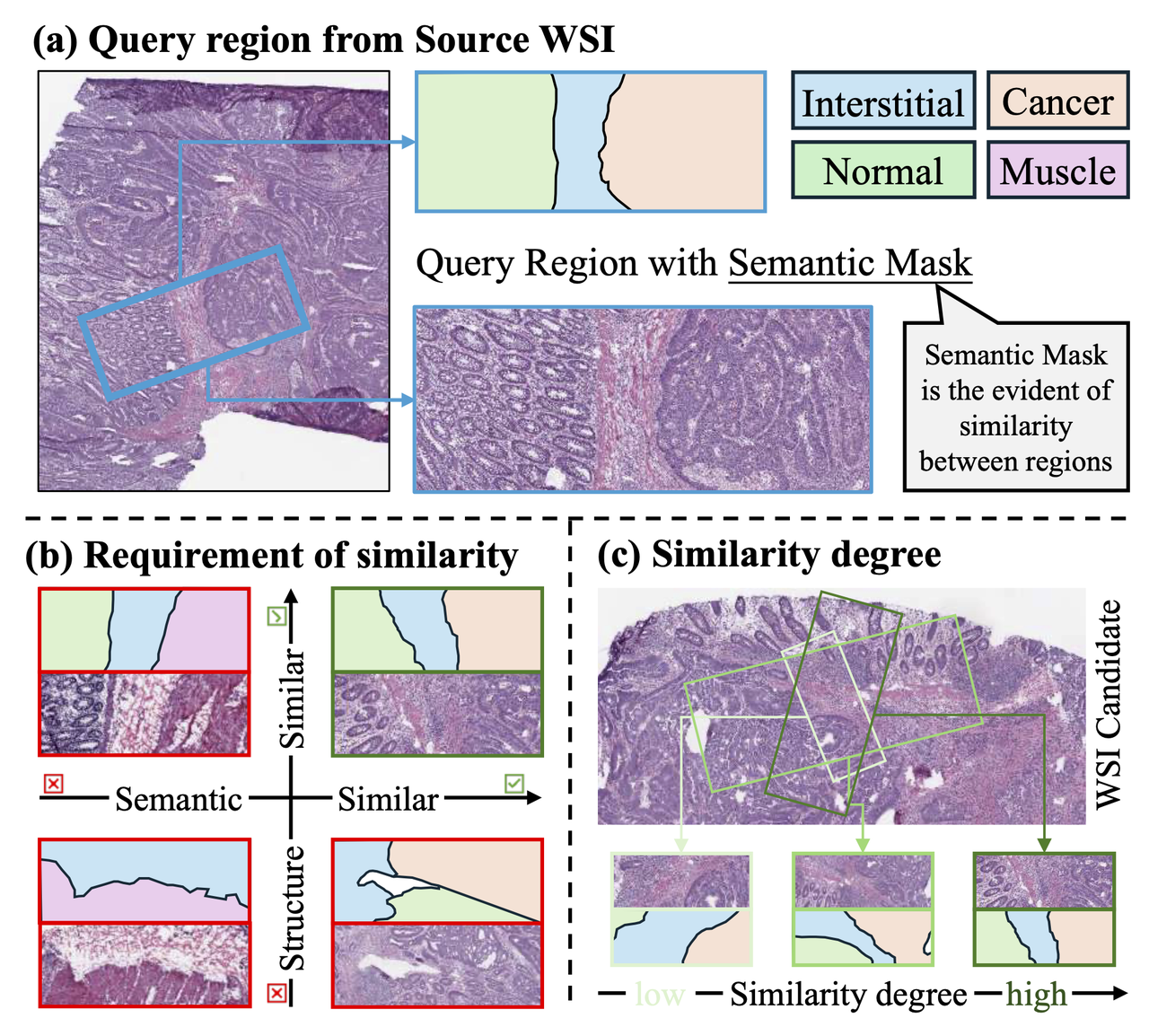
URICA:病理切片检索系统
针对病理全景切片(WSI)检索中的区域级特征表示难题,我们提出了统一区域仿射识别检索算法(URICA),通过语义镶嵌与亲和一致性机制实现旋转与尺度不变的精确区域匹配,并在理论上证明其可近似像素级掩膜检索。
 粟日
粟日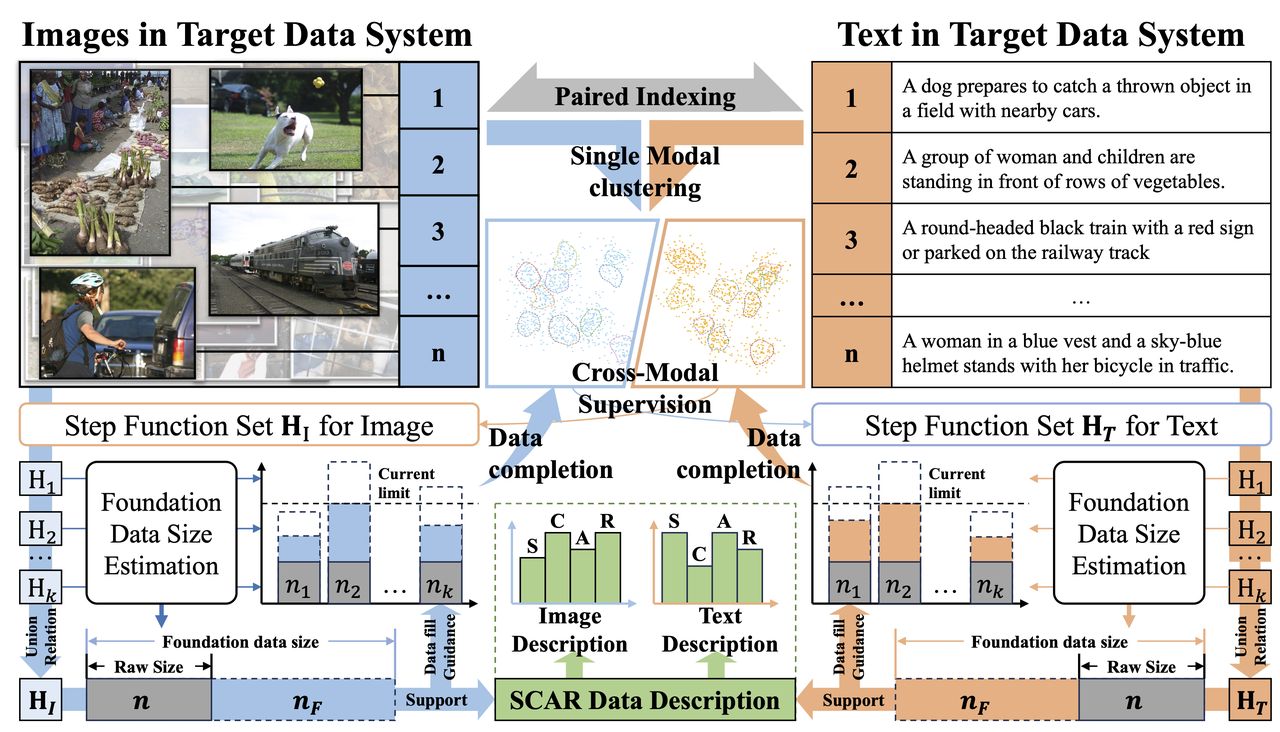
SCAR:一种通用的多模态数据集质量评测
本研究结合数据中心化实践与学习理论,提出以跨模态信号监督为基础的数据质量评估框架。我们定义了基础数据规模(FDS)以刻画模型泛化所需的最小数据量,并提出四维度指标体系 SCAR,用于统一衡量单模态与多模态数据的质量与效用。
 粟日
粟日
基于语义检索的 PubMed 生物医学文献语义检索系统
该项目基于语义检索技术构建了一个面向生物医学研究的 PubMed 论文语义检索系统,旨在实现对海量科研文献的精准语义匹配与高效召回。系统共收录 1,336,133 篇 PubMed 论文,通过将文献内容按 2,000 字符 切分形成语义块,实现了更细粒度的语义索引。最终生成 23,715,917 个待检索节点(chunks),为深层语义检索与科研问答任务提供了坚实的数据基础。
 粟日
粟日 张成
张成